阅读:0
听报道
编者按:
毫无疑问,基因编辑是当前最为火热的一个研究领域。这篇文章解释了时下最为欢迎的基因编辑工具 CRISPR 的工作机制,并解释了单碱基编辑器的出现为何是基因编辑领域的一个突破。
而对于单碱基编辑器来说,脱氨基酶决定其安全性和适用范围。最近,仇子龙及其团队从一种比恐龙还要古老的寄生生物身上得到灵感,通过筛选多样化的胞嘧啶脱氨基酶,构建出一系列新的胞嘧啶碱基编辑器(CBEs),使其安全性和适用范围得到了优化和拓展。
撰文 | 程田林
写在前面的话:DNA与基因
要了解基因编辑,需要对DNA与基因有充分的认识。我们装修自己的房屋,需要有设计图纸,根据图纸来按部就班地完成装修。形象一点而言,DNA就是人体的设计图纸,人体怎样搭建,全靠DNA指引。绘制房屋设计图纸,最基本的便是两种线条:直线,弧线;人体的设计图纸DNA最基本的则是四种碱基:ATGC。房屋设计图纸中,通过线条的组合,我们可以描绘出书房、厨房、卧室、餐厅等等;通过碱基的组合,我们可以设计出搭建人体所需要的各种蛋白质及RNA,对应着蛋白质及RNA的碱基组合便是基因;基因描绘的,其实就是人体的“书房”、“厨房”、“卧室”、“餐厅”等等。房屋设计图纸出错,我们可以通过橡皮或是修正液加以改正。基因出了错,我们该如何改正?基因编辑工具就是负责修正基因的工具。为了改正房屋设计图纸,我们希望能有更好用的橡皮或修正液,最好能安全无痕;我们不断地开发优化基因编辑工具,当然也是希望能安全无痕的改正出错的基因。
一、CRISPR-Cas9系统的诞生
生物学研究可以看做是 “寻宝” 之旅,每名研究者都在寻找自己眼中的 “宝藏”。很多时候,预期的 “宝藏” 未能寻到,却在不经意间开启了 “藏宝洞”。CRISPR-Cas9 系统的发现就是这样的意外。
1987年,大阪大学的 Yoshizumi Ishino 及其同事正关注于大肠杆菌的iap基因,他们已经完成了该基因的测序工作。按照莫诺&雅各布的乳糖操纵子理论,基因的上下游很可能有调控元件的存在(图1A)。于是,Yoshizumi Ishino 也就顺理成章地对iap基因的上下游序列进行了测序。只是,他们没有寻找到预期的 “宝藏”——调控元件,而是发现了无法破解的密码——五段重复的DNA序列。想象一下跨栏比赛的栏杆,研究者发现了五个栏杆(重复的DNA序列),栏杆之间是长度相同但序列不同的DNA片段(图1B)。这种特征的序列是前无古人的,研究者很懵。当然,他们还是写了文章,描述了这一奇怪的发现,但没人知道这意味着什么 [1]。
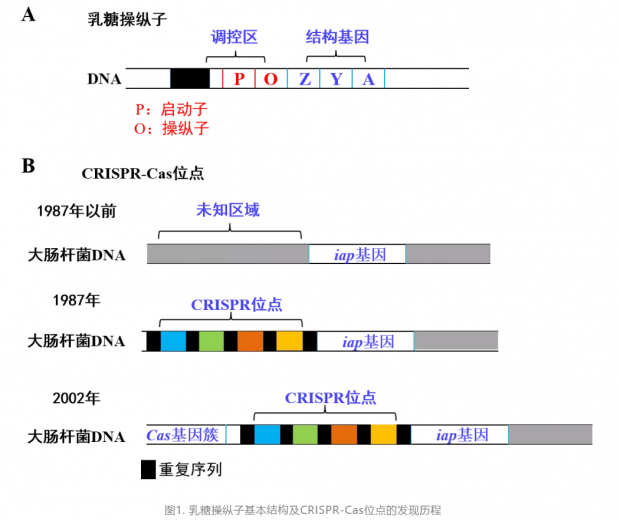
到了二十世纪九十年代,测序技术不断进步,研究者们在微生物的基因组中不断发现类似的特征序列,当然,依旧无人知晓这些序列的秘密。2002年,乌得勒支大学的 Ruud Jansen 团队决定重新分析这类序列,看看是否能“挖宝”。
首先,这类序列有了名字,叫做 “规律成簇的间隔短回文重复”(Clustered regularly interspaced short palindromic repeats,CRISPR)。CRISPR 的名号自此而始。他们还发现,CRISPR 序列可以转录成 RNA;而且序列附近似乎总是有类似的基因相伴,于是这些基因就叫做Cas(CRISPR-associasted),Cas编码的蛋白能切割 DNA(图2)[2]。至此,CRISPR-Cas 系统成型,但它存在的意义依然是一片空白。
后来,研究者发现,CRISPR 序列跟某些噬菌体(这是一种感染细菌的病毒)基因组的部分序列很匹配。至此,细菌与噬菌体被联系在了一起。此时,进化生物学家 Eugene Koonin入场:他整合了关于 CRISPR-Cas 系统的所有碎片化信息,指出,CRISPR-Cas 系统是细菌的免疫系统,是细菌对抗病毒的武器 [3]。
进化生物学家做完预测就已经完成任务。不过听者有意。微生物学家Rodolphe Barrangou 很喜欢这个理论,就去设计实验加以验证。结果发现 Koonin 的理论竟然是对的!也就是说,被噬菌体感染后,细菌会通过 CRISPR-Cas 系统切割噬菌体的基因组以保留证据;如果细菌能侥幸存活,面对同样的噬菌体感染时,它们就能利用已有的证据消灭噬菌体以保护自己 [4]。
此时,CRISPR-Cas 系统的功能基本确定,但具体的细节依然有待研究。此时关键人物 Jennifer Doudna 与 Emmanuelle Charpentier 入场,正是她们的研究让我们明白了 CRISPR-Cas 系统抵御外敌入侵的详细机制,也让基因编辑成为了可能。具体而言,这一机制可分为三步:
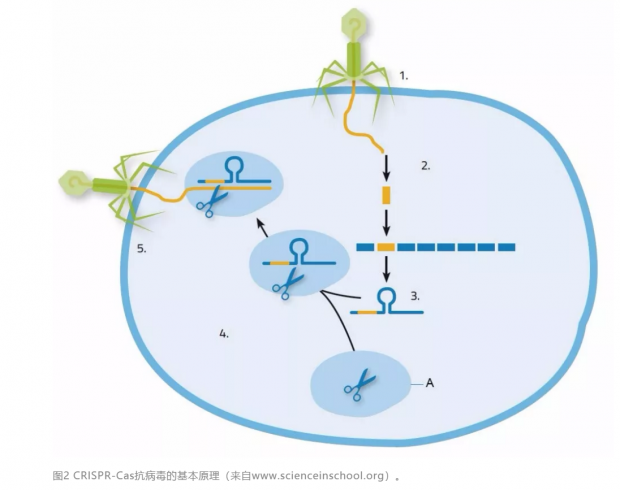
第一步是Cas蛋白复合物对外敌DNA的识别,保留证据(长度为30-50bp的外敌DNA(被称作原型间隔序列))在自己的基因组(CRISPR位点)中(图2,步骤1-2)。
第二步是 CRISPR 位点的转录,最终形成两条短链的 crRNAs(CRISPR来源的RNAs)和 tracerRNA(反式作用的crRNA)。其中 crRNAs 负责携带外敌信息,这是 CRISPR-Cas 系统识别并对抗外敌入侵的基础(图2,步骤3)。
第三步是清除入侵的外敌 DNA。如果 CRISPR-Cas 系统是武器,前两步好比武器零件的制造,这一步就是武器的组装:Cas9 蛋白、crRNA 和 tracerRNA 组成整体,其中 crRNA 好比瞄准镜,精确制导,Cas9 蛋白则像一把剪刀,去剪断被锁定的外敌 DNA(图2,步骤4-5)。[5-6]
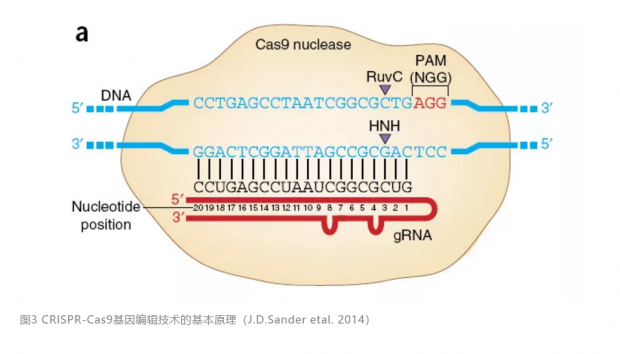
大家都欣喜若狂!CRISPR-Cas 系统能清除外敌 DNA,是因为 crRNA 的序列与外敌 DNA 序列匹配。如果改变瞄准镜 crRNA 的序列,使之匹配其它物种的基因组序列,岂不是指哪打哪?
经过不断的优化,指哪打哪的 CRISPR-Cas9 武器真的诞生了(图3)[7]。
二、DNA单碱基编辑器的诞生
最开始,CRISPR-Cas9 是剪刀,剪断 crRNA 瞄准的基因组位点。但是,两把剪刀不嫌多,有三把更好。研究者们各显神通,“魔改” 系统,将 CRISPR-Cas9 系统打造成了十八般武器:比如,剪刀太锋利,就把剪刀磨钝,只让 CRISPR-Cas9 系统负责定位或是在基因组上留下缺口,而不要完全切断基因组;要想调控基因的转录,就给钝剪刀加上转录调节器;要想精修基因组,就给钝剪刀加上各样的修剪器。
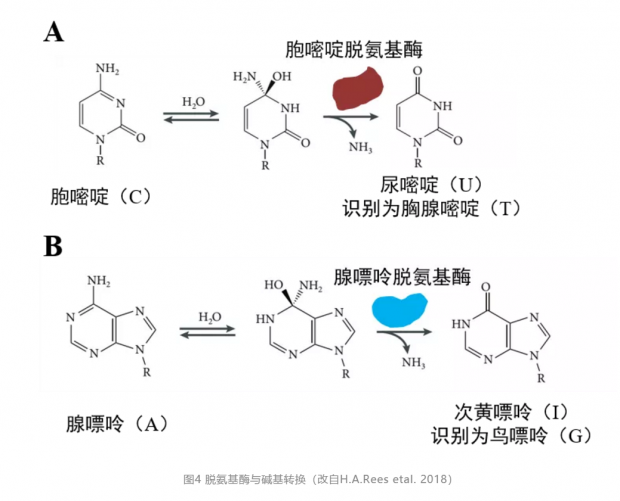
我们都知道,DNA 序列是由四种不同的碱基组成,通过不同的排布,便可以储藏丰富的信息。如果有特定的碱基改变,其储藏的信息也会发生改变。那么,CRISPR-Cas9 系统能否被改造成精准改变特定碱基的武器?细胞内有一类酶,叫做碱基脱氨基酶,其中有两种,一是胞嘧啶脱氨基酶,它可以让胞嘧啶(C)变成尿嘧啶(U),而U在基因组中会被替换成胸腺嘧啶(T),从而能实现碱基的改变(C->T)(图4A);另一类是腺嘌呤脱氨基酶,它可以让腺嘌呤(A)变成次黄嘌呤(I),在基因组中I发挥鸟嘌呤(G)的功能,从而能实现碱基的改变(A->G)(图4B)。
而将磨钝的 CRISPR-Cas9 系统与脱氨基酶组合,研究者获得了可实现碱基转换的新武器——单碱基编辑器(Baseeditors,BEs)。介导 C->T 改变的被称为胞嘧啶碱基编辑器(CBEs)[8],而介导A->G改变的被称为腺嘌呤碱基编辑器(ABEs)[9]。不过单碱基编辑器有两大问题,一是瞄准镜的范围小,有的位置瞄不到;二是有脱靶,不小心会改变其它位置的碱基,从而引发安全风险 [10]。
三、单碱基编辑器的深度演化
要改造优化单碱基编辑器,更换不同的脱氨基酶是可行的策咯。CBE 系统中常用的脱氨基酶分别来自大鼠的 APOBEC1,人的 AID1 和七鳃鳗的 PmCDA1,它们有着相似的编辑范围。自然界是否还有另类的脱氨基酶存在?这需要深入挖掘。
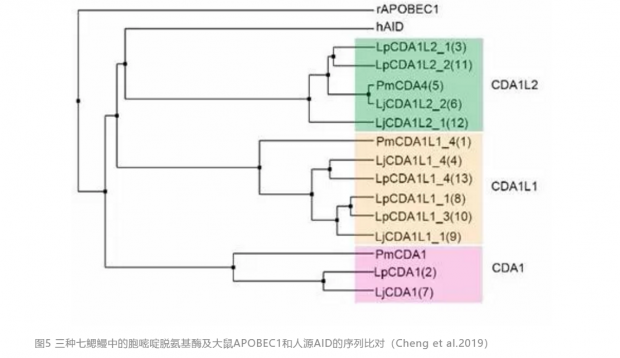
在挖矿一般的查找文献和数据库后,中国科学院脑科学与智能技术卓越创新中心研究员仇子龙及其团队发现,七鳃鳗中有着相当多样的胞嘧啶脱氨基酶(图5)[11],值得深入探索。
七鳃鳗,是一种比恐龙还要古老的寄生生物,它们在地球上的生存时间已经超过3.6亿年。作为寄生生物,七鳃鳗有吸血狂魔的恶名(图6)。在美食家眼中,七鳃鳗却又享有盛名:它的美味曾经让英王亨利一世无法自拔,并最终吃的太多,腹胀而死。
这样一种 “史前” 美味,给单碱基编辑器的优化带来了希望。
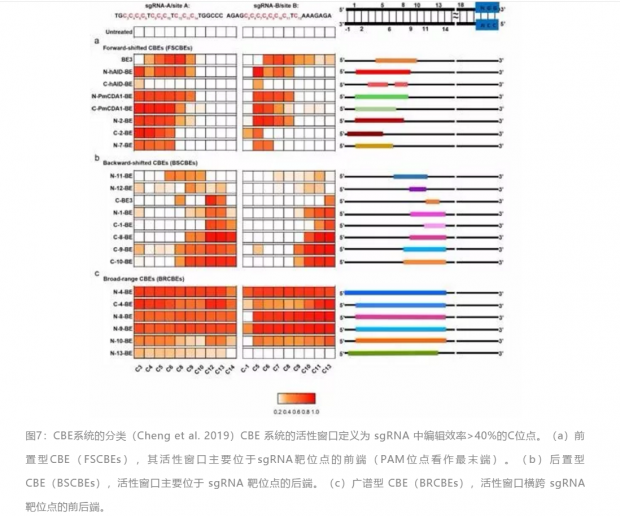
七鳃鳗会表达多种不同的胞嘧啶脱氨基酶,而且不同种的七鳃鳗所含的酶功能各有差异 [11]。这些多样化的酶与 CRISPR/Cas 系统组合后极大的拓展了 CBEs 的活性范围,这让瞄准范围更广,极大地丰富了单碱基编辑的工具箱。如同乐高积木一样,通过不同的组合策略,我们获得了一系列的 CBEs,它们有着不同特色的瞄准镜,使得活性范围更加多样化,从而有着不同的适用场景,这让工具箱中的CBE 工具更加丰富,在使用上也有了更大的选择余地(图7)[12]。此外,新添加的 CBE 工具在安全性上也有了明显的改善,这为单碱基编辑技术的应用提供了更多选择,新的胞苷脱氨酶也为未来单碱基编辑工具的开发和改善提供了基础。
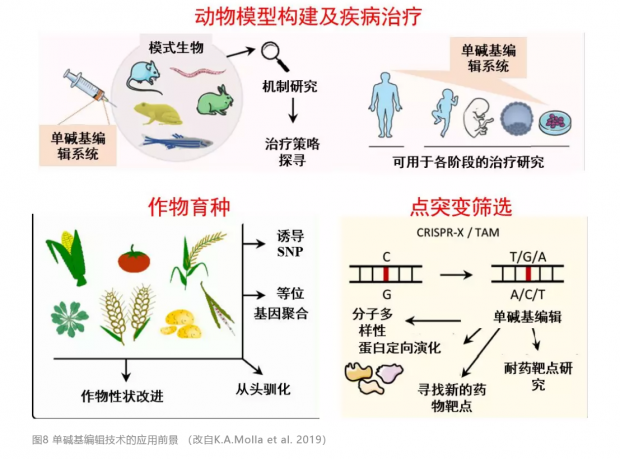
单碱基编辑技术有着相当广泛的应用(图8),它在动物模型的构建、疾病治疗、作物育种以及点突变筛选等等多个方面都极具潜力 [13]。开发安全性高、适用范围广的单碱基编辑工具将有助于其潜力的兑现,从而更好的服务于生物医学研究。新的 CBE 工具在安全性和适用范围上均取得了相当大的进步,为单碱基编辑技术的进步做出了贡献。
作者简介
程田林博士,助理研究员,中国科学院脑科学与智能技术卓越创新中心,中国科学院神经科学研究所
参考文献:
1. Ishino, Y., Shinagawa, H., Makino, K., Amemura, M., and Nakata, A. (1987).Nucleotide sequence of the iap gene, responsible for alkaline phosphataseisozyme conversion in Escherichia coli, and identification of the gene product.J Bacteriol 169, 5429-5433.
2. Jansen, R., Embden, J.D., Gaastra, W., and Schouls, L.M. (2002). Identificationof genes that are associated with DNA repeats in prokaryotes. Mol Microbiol 43, 1565-1575.
3. Makarova, K.S., Grishin, N.V., Shabalina, S.A., Wolf, Y.I., and Koonin, E.V.(2006). A putative RNA-interference-based immune system in prokaryotes:computational analysis of the predicted enzymatic machinery, functionalanalogies with eukaryotic RNAi, and hypothetical mechanisms of action. BiolDirect 1, 7.
4. Marraffini, L.A. (2015). CRISPR-Cas immunity in prokaryotes.Nature 526, 55-61.
5. Doudna, J.A., and Charpentier, E. (2014). Genome editing. The new frontier ofgenome engineering with CRISPR-Cas9. Science346, 1258096.
6. Hsu, P.D., Lander, E.S., and Zhang, F. (2014). Development and applications ofCRISPR-Cas9 for genome engineering. Cell157, 1262-1278.
7. Sander, J.D., and Joung, J.K. (2014). CRISPR-Cas systems for editing,regulating and targeting genomes. Nat Biotechnol 32, 347-355.
8. Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A., and Liu, D.R. (2016).Programmable editing of a target base in genomic DNA without double-strandedDNA cleavage. Nature 533, 420-424.
9. Gaudelli, N.M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson,D.I., and Liu, D.R. (2017). Programmable base editing of A*T to G*C in genomicDNA without DNA cleavage. Nature 551,464-471.
10. Rees, H.A., and Liu, D.R. (2018). Base editing: precision chemistry on thegenome and transcriptome of living cells. Nat Rev Genet.
11. Holland, S.J.,Berghuis, L.M., King, J.J., Iyer, L.M., Sikora, K., Fifield, H., Peter, S.,Quinlan, E.M., Sugahara, F., Shingate, P.,et al. (2018). Expansions, diversification, and interindividual copy numbervariations of AID/APOBEC family cytidine deaminase genes in lampreys. Proc NatlAcad Sci U S A 115, E3211-E3220.
12. Cheng, T.-L., Li, S., Yuan, B., Wang, X., Zhou, W., and Qiu, Z. (2019).Expanding C–T base editing toolkit with diversified cytidine deaminases. NatureCommunications 10, 3612.
13. Molla, K.A., andYang, Y. (2019). CRISPR/Cas-Mediated Base Editing: Technical Considerations andPractical Applications. Trends Biotechnol.

话题:
0
推荐
财新博客版权声明:财新博客所发布文章及图片之版权属博主本人及/或相关权利人所有,未经博主及/或相关权利人单独授权,任何网站、平面媒体不得予以转载。财新网对相关媒体的网站信息内容转载授权并不包括财新博客的文章及图片。博客文章均为作者个人观点,不代表财新网的立场和观点。



 京公网安备 11010502034662号
京公网安备 11010502034662号 